Teoretisk biokemi
Kort beskrivning
Leif Erikssons forskargrupp använder avancerade datorsimuleringar för att utveckla nya läkemedel eller beskriva olika proteiners struktur, funktion och växelverkan. Till sin hjälp har de olika superdatorkluster i Sverige. Gruppens forskning fokuserar mycket på att identifiera nya molekyler för att behandla cancer, exempelvis genom att blockera cellernas förmåga att skydda sig mot stress. Gruppen utvecklar även modeller av proteinkomplex och studerar hur cellerna läser DNA (så kallad transkription).
Vår forskning
I vår forskning använder vi avancerade beräkningskemiska och bioinformatik-baserade verktyg (homologimodellering, protein-protein dockning, virtuell screening, molekyldynamik-simuleringar och QM/MM), för att utveckla nya substanser som kan fungera som läkemedel eller för att bättre förstå proteiners växelverkan och funktion.
Mycket av gruppens forskning fokuserar på behandling av cancer, och vi studerar flera olika måltavlor i detta. Bland annat har vi under senare år arbetat mycket med proteinet IRE1 (inositol requiring enzyme 1) som är involverat i cellers svar på/skydd mot stress (unfolded protein response; UPR). Vi studerar bland annat hur olika protein bildar komplex med IRE1 i ’UPRosomen’, samt har utvecklat den första BBB-penetrerande inhibitorn mot IRE1’s kinas-säte. Denna substans har vi med stor framgång testat i djurexperiment för behandling av den dödliga hjärntumören glioblastoma multiforme (GBM).
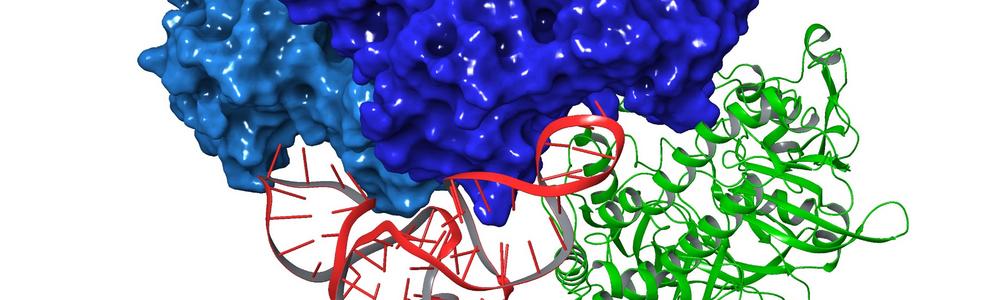
Förutom IRE1 och en rad andra måltavlor inom cancerbehandling arbetar vi även med ny metodik för bättre prediktion av protein-komplex, samt maskinlärning inom läkemedelsutveckling. En del av teamet forskar också om transkriptionsfaktorer och hur dessa känner av ’rätt’ sekvens av DNA, samt hur DNA’s flexibilitet styrs av den specifika ordningen på nukleinsyror.
Artificiell intelligens och maskininlärning
Vår forskargrupp har utvecklat två datorprogram som bygger på avancerad maskininlärning (ML).
Vårt program i-TripleD kan extremt snabbt och med oöverträffad noggrannhet bestämma liganders dissociationskonstanter eller generera helt nya läkemedelskandidater. Nyligen screenades en databas på över 1 miljard substanser på mindre än 36 timmar; en process som med vanlig datorbaserad screening skulle ha tagit fem månader.
Vår andra ML-baserade mjukvara är programmet iPIN, med vilket vi med nära nog 100% noggranhet kan bestämma vilka proteiner som växelverkar med varandra.
Metoderna är nu föremål för patentering och har i samarbete med Chalmers Ventures och Chalmers Entreprenörskola lett till att avknoppningsföretaget ANYO Labs bildades. Ytterligare metoder inom ML-baserad metodik är under utveckling.



